9 Quantificação
Após a etapa de extração, determinar a concentração de DNA, assim como a sua qualidade e pureza, são fatores importantes para o desenvolvimento de estudos em biologia molecular. Dessa maneira, as amostras obtidas podem ser avaliadas quanto à sua concentração e qualidade. Essa avaliação pode acontecer de algumas maneiras, como realizando uma eletroforese em gel de agarose, através da medição da absorbância com o uso de espectrofotômetros ou utilizando fluorímetro por meio das alterações nas características de fluorescência na presença de DNA.
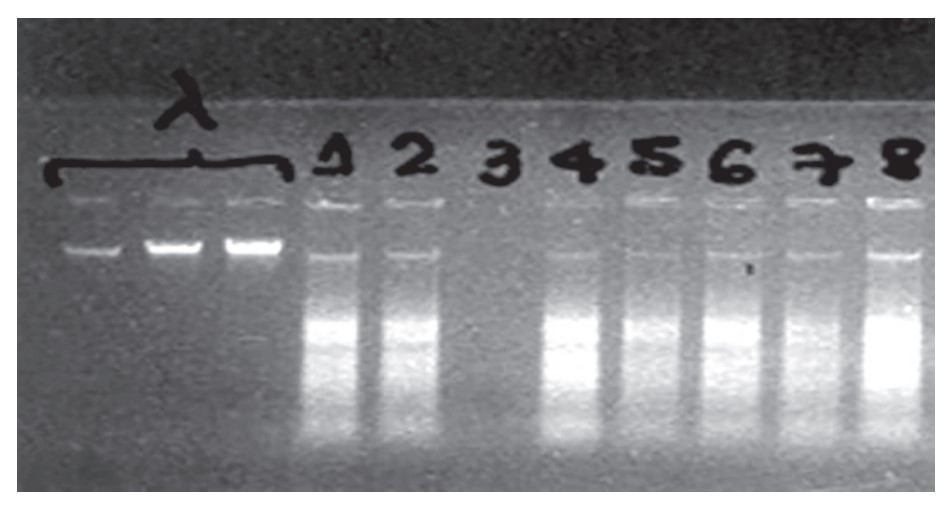
A quantificação irá fornecer informações sobre a concentração de DNA extraído, assim como a integridade das amostras que serão trabalhadas. Na quantificação utilizando gel de agarose, um gel com concentração de 1% é preparado e as amostras de DNA são aplicadas juntamente com amostras de concentrações conhecidas. Para essa finalidade, o DNA do bacteriófago Lambda é comumente utilizado como padrão. O gel é normalmente submetido a eletroforese e é realizada uma estimativa visual através da comparação com o DNA padrão para determinar a concentração de DNA em cada amostra, analisando a fluorescência e espessura das bandas, obtendo valores aproximados. Além disso, as amostras consideradas de boa qualidade irão formar bandas íntegras, enquanto amostras degradadas ou com presença de moléculas de RNA apresentarão rastros ao longo do gel.
Por outro lado, a quantificação em espectrofotômetros, como por exemplo o NanoDrop, fornece dados um pouco mais exatos sobre a concentração de DNA presente nas amostras. Entretanto, ainda são valores baseados em um calibrante, tomado como padrão, além de ser necessário adquirir aparelhos específicos para realizar a quantificação do DNA, tendo um custo elevado quando comparado à quantificação em gel. Essa quantificação é baseada na medição da quantidade de luz absorvida pelo DNA em solução no comprimento de onda de 260 nm. Quanto maior for a absorção de luz nesse comprimento de onda, maior a concentração de DNA extraído na amostra.
Já o fluorímetro é um dos métodos mais sensíveis e geralmente utilizado para amostras com pouca concentração de DNA ou que tenham contaminantes que absorvem comprimentos de onda de 260 nm. Essa análise acontece pela luz emitida de moléculas fluorogênicas, sendo um método alternativo para avaliar a concentração de DNA e RNA marcando a amostra com um marcador fluorescente (corante fluorescente). Porém, também é um método com um custo elevado e demanda um tempo maior para preparação das amostras.
A quantificação irá mostrar se as amostras de DNA extraídas precisam ser diluídas para a realização da PCR, pois é preciso ter uma concentração ideal. Dessa forma, quando as amostras forem quantificadas com valores superiores a 5 ng/μL, elas precisam ser diluídas. A fórmula utilizada para isso é \(C1 * V1 = C2 * V2\), onde serão encontradas as quantidades de DNA e água Milli-Q necessárias para que o DNA fique na concentração final de 5 ng/μL.
9.1 Quantificação com gel de agarose
Gel agarose 1%.
Correr por aproximadamente 40 minutos, a 100 volts.
DNA lambda (pipetar 5 μL + 1 μL de GelRed em cada um):
5 ng/μL – 25 ng
10 ng/μL – 50 ng
20 ng/μL – 100 ng
GelRed
2 μL de DNA + 1 μL de loading + 1 μL de GelRed + 1 μL de água Milli-Q*
*água Milli-Q é opcional
Safer
3 μL de DNA + 1 μL de Safer + 2 μL de água Milli-Q
9.2 Quantificação com NanoDrop
Fica localizado no DBC (Bloco H67 – Pegar a chave no laboratório 17);
Levar para quantificação:
Amostras que serão quantificadas;
Pipeta de 20 uL;
Ponteiras amarelas;
Luvas;
Jaleco;
Caneta e caderno para anotações;
Tampão TE;
Água Milli-Q;
Alíquota dos tampões de extração (Rehidratation Solution e Tampão AE – dependendo de qual kit foi utilizado para extração do DNA).
Após pegar a chave e entrar na sala, o Nanodrop fica localizado após a segunda porta;
O equipamento deve ser ligado na tomada (110V);
Após ligado, certifique-se de que a local onde irão ser colocadas as amostras esteja limpo. Caso contrário, limpe-a cuidadosamente com água Milli-Q;
Selecionar a opção “dsDNA”;
Com a opção “dsDNA” selecionada, o Nanodrop irá pedir para dar um “Blank” (branco no equipamento). Deve ser pipetado 2 uL da solução tampão usada para armazenamento do DNA na etapa final de extração. Esse processo deverá ser feito duas vezes (o próprio equipamento irá pedir);
Lembrar sempre de limpar o local onde são pipetadas as amostras com o papel higiênico que fica junto com o equipamento. Principalmente entre cada amostra a ser quantificada para não haver contaminações;
Antes de começar a quantificação com as amostras, quantificar 2 uL de água Milli-Q para conferir o valor (geralmente fica próximo de -2);
Após isso, as amostras podem ser quantificadas. Agite suavemente a amostra para garantir homogeneidade;
Pipetar sempre 2 uL de cada amostra na área de leitura do Nanodrop, fechar a tampa do equipamento cuidadosamente, apertar o botão“Measure” e aguardar a conclusão da leitura;
O valor irá aparecer na tela e deve ser anotado. O Nanodrop fornecerá um espectro de absorção e valores de concentração para a amostra. Observar os valores de A260 (absorbância a 260 nm), que é usado para calcular a concentração de DNA. A relação A260/A280 também é importante para avaliar a pureza da amostra. Uma relação ideal é geralmente em torno de 1.8 para DNA puro;
Para a próxima amostra, a tampa do equipamento deve ser aberta, o local limpo com o papel e só após isso a próxima amostra deve ser pipetada, repetindo sempre esses processos;
Após finalizar todas as quantificações, limpe a área de leitura com água Milli-Q, fazendo uma leitura final para remover qualquer resíduo da amostra anterior. Secar delicadamente com o papel e fechar a tampa do Nanodrop, tirando-o da tomada;
Dicas:
Sempre use água Milli-Q para preparar suas amostras. Caso algum valor dê diferente do esperado (por exemplo, valores negativos), faça uma quantificação da água novamente.
Evite bolhas de ar na área de leitura do Nanodrop, pois elas podem afetar a precisão da leitura.
Calibre o Nanodrop usando água Milli-Q antes de cada sessão de medição. Para finalizar a utilização, repita esse procedimento.
Lembre-se de consultar o manual específico do seu Nanodrop e seguir os protocolos e recomendações fornecidos pelo fabricante.
9.3 Diluição do DNA para 5ng/μL
\(C1 * V1 = C2 * V2\)
C1 – concentração da amostra (inicial)
V1 – quantidade de DNA necessária (descobrir)
C2 – concentração para diluir (vai diluir para 5 ng)
V2 – volume final total (50 μL)
Obs: Quantidade de água Milli-Q é: 50 μL - quantidade de DNA